Symbiologik : jouez avec les gènes
Cet article est devenu obsolète, de par son contenu ou sa forme, il est donc archivé.
l'assemblage de fragments d'ADN,
la recherche de motifs communs à plusieurs protéines,
la construction d'arbres phylogénétiques.
Dans la réalité, ces tâches sont exécutées par ordinateur sur des volumes de données énormes. En effectuant ces tâches « à la main », sur des exemples simplifiés, vous vous ferez une idée des méthodes informatiques mises en œuvre par les bioinformaticiens. Le temps que vous allez y passer vous fera comprendre pourquoi il a été indispensable d’automatiser ces tâches.
 |
Ce jeu en Flash nécessite un plug-in Adobe Flash.
Pour comprendre le but du jeu : quelques notions de biologie moléculaire
Jeu 1
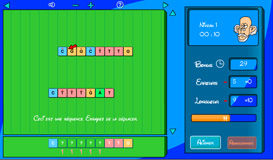
Le premier jeu consiste à assembler des fragments d’ADN. Quelle est l’utilité ? En fait, c’est une phase incontournable dans le séquençage des génomes ; celui-ci consiste à traduire les longues molécules d’ADN en un texte à 4 lettres : A, C, G et T, qui sont les initiales des nucléotides qui s’enchaînent le long de cette molécule. Les machines qui séquencent l’ADN (les séquenceurs) ne sont pas capables de décoder directement ces longues séquences. Au maximum, seuls des morceaux de 500 à 1 000 caractères peuvent être traités.
Le séquençage commence donc par un découpage aléatoire de la molécule d’ADN en milliers (voire millions) de petits fragments. Ces fragments se chevauchent, mais la position des uns par rapport aux autres est perdue. Ils sont ensuite décodés indépendamment par les séquenceurs. La phase finale consiste à reconstituer le texte sur la base de ces petits fragments. On procède par étapes successives : deux fragments qui se chevauchent donnent naissance à un fragment plus important, et ainsi de suite, jusqu’à l’obtention d’un fragment unique qui représente le texte du génome. Cela s’apparente à la constitution d’un puzzle de plusieurs millions de pièces.
Le jeu illustre cette reconstruction. Plusieurs niveaux de difficulté sont proposés. Ils traduisent la complexité de la tâche. En réalité, les séquenceurs ne sont pas parfaits. Ils font des erreurs que les méthodes informatiques doivent compenser.
Jeu 2
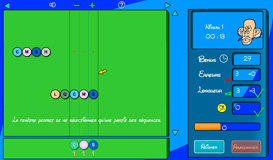
Le but du deuxième jeu est de rechercher des motifs communs à plusieurs protéines. Ces dernières jouent un rôle essentiel dans la cellule. Elles ont de multiples fonctions, principalement déterminées par leur conformation spatiale. On peut les représenter comme un chapelet d’acides aminés (les perles) replié sur lui-même. L’activité est souvent liée à quelques acides aminés qui ont une disposition bien déterminée dans l’espace et au sein de la protéine.
Cet agencement particulier de quelques acides aminés (un motif) constitue une signature de la fonction. Lorsqu’il est reconnu dans leur séquence, il sert à affecter une fonction à de nouvelles protéines. Inversement, ayant déterminé expérimentalement qu’un groupe de protéines possède des fonctionalités semblables, on peut rechercher le ou les motifs responsables de cette activité.
Le jeu est orienté vers la découverte de motifs. Etant donné un ensemble de protéines, il faut rechercher les motifs communs. En réalité, l’ingénierie du vivant conduit à des situations où l’extraction d’un motif se révèle une opération délicate. En partant de cas simples, le jeu se complexifie petit à petit pour pointer progressivement sur les réelles difficultés à faire émerger un motif satisfaisant. Bonne recherche !
Jeu 3
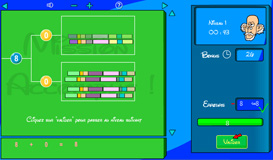
Le troisième jeu vous invite à construire des arbres phylogénétiques. Ces entités visualisent l’évolution des organismes à partir de la structure de leurs gènes. Au cours des millénaires, des millions d’espèces se sont créées, donnant progressivement naissance à des milliers de gènes différents. Mais malgré leur différence, les gènes gardent en mémoire cette histoire évolutive.
Schématiquement, un gène est composé d’un assemblage de modules ou régions. Entre deux gènes, certains modules ont été modifiés ou perdus ; d’autres ont été acquis ou dupliqués. En analysant les différences entre gènes, on peut se faire une idée de la proximité d’un ancêtre commun.
À partir d’un ensemble de gènes composés de plusieurs modules, le jeu consiste à les regrouper par familles. Une famille comprend des gènes proches, c’est-à-dire des gènes qui ne comportent que très peu de modules différents. Récursivement, on peut ensuite regrouper les familles pour construire progressivement l’arbre phylogénétique, ou plus simplement l’histoire de ces gènes.
Voir aussi les autres documents de la thématique Bioinformatique, et en particulier :
- sur les génomes et leur séquençage, Vous n’auriez pas vu un gène, par hasard ?
- sur l’étude des protéines, La post-génomique ou les mystères de la cellule et La bioinformatique en protéomique : analyse des spectres de masse
- sur la comparaison et l’alignement de séquences, Alignement optimal et comparaison de séquences génomiques et protéiques.
Newsletter
Le responsable de ce traitement est Inria. En saisissant votre adresse mail, vous consentez à recevoir chaque mois une sélection d'articles et à ce que vos données soient collectées et stockées comme décrit dans notre politique de confidentialité
Niveau de lecture
Aidez-nous à évaluer le niveau de lecture de ce document.
Votre choix a été pris en compte. Merci d'avoir estimé le niveau de ce document !

Nicolas Deroche
