Stocker les données : la piste prometteuse de l’ADN
La croissance des données numériques
Chaque jour, l’humanité produit une masse de données qui, d’un point de vue stockage de l’information, représente l’équivalent de plusieurs milliards de disques durs. Les réseaux sociaux, l’Internet des objets, le développement de la 5G, sont des exemples typiques d’applications numériques qui participent activement à cet amoncellement de données et qui, de plus, ne cessent de croître avec le temps. La figure 1 illustre cette progression avec une projection sur la prochaine décennie.

Figure 1 : Estimation du volume de données numériques créées par an dans le monde, en Zettaoctets. Un Zettaoctet (Zo) équivaut à mille milliards de gigaoctets.
L’impact de cette production exponentielle de données sur le stockage est immédiat et représente un enjeu de taille. Les données sont actuellement concentrées dans de gigantesques infrastructures (appelées centres de données ou data centers) dont les plus grandes ont une capacité de stockage de plusieurs exaoctets (soient 1018 octets équivalant à 1 milliard de gigaoctets), ce qui correspond, pour chacun, à une fraction significative de la production journalière mondiale. Cette stratégie de stockage qui nécessite d’investir constamment dans de nouveaux data centers pointe ses limites, que ce soit du point de vue de l’économie, de l’environnement, de la cybersécurité, de la souveraineté ou encore du patrimoine numérique.
Le stockage digital
Le stockage de ces volumes d’informations, tel qu’il s’opère actuellement, n’est pas sans conséquences. En janvier 2021 on comptait près de 8 000 « data centers » dans le monde. Avec une croissance estimée de 20 % chaque année, leur nombre double tous les quatre ans, tout comme la quantité d’énergie nécessaire pour alimenter leurs milliers de serveurs informatiques.
En effet, le fonctionnement des data centers entraîne des coûts économiques et une empreinte environnementale considérables : un data center coûte environ 1 milliard de dollars sur dix ans (ce qui représente une dépense globale de 8 millions de milliards de dollars aujourd’hui), consomme environ 100 mégawatts/heure (soit 10 % d’une centrale nucléaire) et occupe une surface au sol d’approximativement 100 000 m2 (l’équivalent de 80 piscines olympiques). On estime donc que d’ici 2030, le stockage et la transmission de nos données représenteront approximativement 20 % de la consommation totale d’énergie.
Le problème est également aggravé par la durée de vie limitée des supports de stockage, qui va de 3 à 5 ans pour les disques durs et de 10 à 20 ans pour les bandes magnétiques. Les centres de données migrent continuellement leurs données, ce qui entraîne un énorme gaspillage de matériel.
Les défis des technologies d’archivage
Depuis longtemps, les bandes magnétiques constituent la meilleure option pour l’archivage à long terme. C’est une technologie mature, bon marché, fiable et sûre. Cependant, l’écriture et la lecture sont des opérations lentes. Contrairement aux disques, l’accès aux fichiers n’est pas aléatoire mais séquentiel : la bande doit être mécaniquement bobinée jusqu’à l’endroit souhaité pour être lue ou écrite.
Mais plus gênant, les formats physiques et logiques se succèdent rapidement. Les fabricants actualisent continuellement leurs matériels et leurs logiciels pour augmenter les capacités de stockage. Et, pour les utilisateurs et utilisatrices, cela implique une mise à jour continuelle des matériels et des logiciels. Cette obsolescence rapide n’est pas sans effet. Par exemple, les contenus audio enregistrés sur des bandes depuis un siècle représentent des millions d’heures de musique, d’émissions de radio, de discours, de débats ou de conférences. Ces enregistrements donnent aux historiens un aperçu unique de notre passé, mais les fabricants ont pratiquement cessé de fabriquer des lecteurs de bandes audio. Cela signe l’arrêt de mort des contenus qui n’ont pas encore été transférés sur des formats plus récents.
Dans ce contexte, il est nécessaire de trouver un meilleur support pour stocker nos données. Un support capable de stocker des quantités massives d’informations pendant des siècles sans coûts économiques, opérationnels et environnementaux, sans risque d’obsolescence et pouvant être facilement conservé en lieu sûr !
L’ADN comme support de stockage
Pour faire simple, on peut considérer deux principales technologies qui ont permis de stocker de l’information pendant des millénaires sans problème d’obsolescence physique : le papier et l’ADN. La civilisation humaine codifie les informations sur papier depuis environ 5 000 ans, mais la nature stocke les informations génétiques dans l’ADN depuis des milliards d’années. Compte tenu des limites auxquelles sont confrontés les supports traditionnels de stockage, l’ADN apparaît comme un matériau plein de promesses.
Tout d’abord, la molécule d’ADN est extrêmement dense : un gramme peut théoriquement contenir 455 exa (1018) octets d’informations, soit l’équivalent de plusieurs milliards de vidéos haute définition (ou HD). Deuxièmement, le stockage sur le long terme ne nécessite pas d’énergie, hormis le contrôle de la température et de l’atmosphère ambiantes. Troisièmement, l’ADN est facile à copier et à consulter : il peut être répliqué par des enzymes spécialisées (polymérases), et il est possible d’accéder sélectivement à un ensemble de brins d’ADN.
Si on prend le seul critère de la densité, le stockage sur ADN se montre extrêmement compétitif par rapport aux supports de stockage usuels (voir la figure 2 ci-dessous). Par contre, comme on le verra par la suite, les temps d’écriture et de lecture sont encore excessivement longs ce qui, pour l’instant, positionne plutôt cette technologie pour l’archivage de données « froides », c’est-à-dire des données qui sont très rarement relues mais pour lesquelles il est essentiel de conserver une copie.

Figure 2 : Densité des différentes technologies de stockage. La densité est exprimée en nombre de bits stockés par mm3. L’échelle est logarithmique pour une meilleure représentation. Une mémoire FLASH (clé USB), par exemple, a une densité de 8 milliards de bits/mm3 (8×109). L’ADN a une densité de stockage 1 million de fois plus importante (8×1015). L’écart avec les autres technologies est sans appel. Figure adaptée de G. Church et al., « Next-Generation Digital Information Storage in DNA », Science, vol 337, issue 6102, 2015.
La molécule d’ADN comme support d’information
La molécule d’ADN possède une structure en double hélice constituée de deux polymères enroulés l’un autour de l’autre. Ces derniers sont constitués d’un enchaînement de quatre nucléotides différents : l’adénine, la thymine, la guanine et la cytosine, symbolisés par les 4 lettres A, T, G et C. Les nucléotides des deux brins d’ADN interagissent entre eux à travers des liaisons hydrogène qui déterminent des règles d’appariement entre nucléotides : l’adénine (A) et la thymine (T) s’apparient au moyen de deux liaisons hydrogène ; la guanine (G) et la cytosine (C) s’apparient au moyen de trois liaisons hydrogène (voir la figure 3 ci-dessous). Ces liaisons sont à l’origine de la configuration en double hélice qui procure à la molécule d’ADN une très grande stabilité au cours du temps.
D’un point de vue informatique, les quatre lettres A, T, G et C constituent un alphabet sur lequel on peut coder de l’information, au même titre que l’alphabet binaire composé uniquement des deux caractères 0 et 1. Un fichier binaire peut être directement transcodé, par exemple, en appliquant les règles suivantes :
| 00 → A | 01 → C | 10 → G | 11 → T |
Ainsi la chaine binaire 00 10 10 11 10 10 10 01 00 01 se transforme en AGGTGGGCAC qui est ensuite convertie en une molécule d’ADN dont un des brins contient exactement cette séquence de nucléotides.
Ici, la molécule d’ADN est considérée comme un simple polymère. Elle n’a pas de fonction biologique particulière et n’est intégrée dans aucun organisme vivant. Elle sert simplement de support au stockage d’information. D’autres types de polymères, peut-être mieux adaptés, pourraient bien sûr être utilisés. L’avantage de l’ADN est que de nombreuses technologies sont disponibles pour le manipuler et continueront de l’être dans le cadre des applications génomiques (santé, agronomie, environnement, etc.).
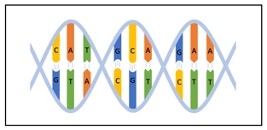
Figure 3 : Molécule d’ADN : elle est structurée en hélice autour de deux brins complémentaires. Chaque brin est un enchaînement de nucléotides A, T, G ou C.
Le cycle de l’écriture et de la lecture de l’ADN
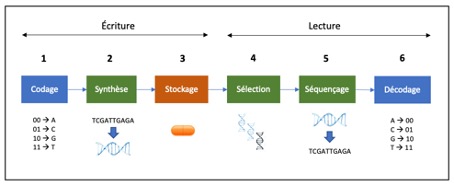
Schématiquement, l’ensemble des opérations pour effectuer le cycle complet « écriture/lecture » se décompose en six grandes étapes, comme illustré par la figure 4 ci-dessous.
Écriture
- Codage : l’information est formatée et transcrite en séquences texte A,C,G,T ;
- Synthèse : le texte A,C,G,T est transformé en molécules d’ADN ;
- Stockage : l’ADN est protégé et conservé sur des supports adéquats.
Lecture
- Sélection : les molécules d’ADN correspondant aux documents à lire sont identifiées ;
- Séquençage : les molécules d’ADN sont converties en séquences A,C,G,T ;
- Décodage : reconstitution des documents à partir du texte A,T,C,G.
Codage de l’information (étape 1)
D’un point de vue traitement du signal, les étapes 2 à 5 peuvent être vues comme un canal de communication bruité. Ces étapes engendrent des erreurs qu’il va falloir prendre en considération pour être en mesure de les corriger à l’étape de décodage. De plus, les molécules d’ADN synthétisées ont des contraintes inhérentes aux technologies de synthèse et de séquençage :
1. Pas de séquences d’homopolymères (suites de nucléotides identiques) pour éviter principalement des erreurs de séquençage. Typiquement, on interdit des suites de plus de 3 ou 4 nucléotides identiques.
2. Un nombre de caractères C,G plus faible que le nombre de caractères A,T. Cette contrainte facilite, par la suite, la faisabilité des diverses opérations de biotechnologie.
3. Pas de motifs répétés dans les séquences pour faciliter le décodage.
Ainsi, pour prendre en compte les erreurs introduites pendant le processus d’écriture et de lecture ainsi que les contraintes relatives au support de stockage, le codage comprend généralement deux phases : le codage source et le codage canal.
Le codage source se charge de formater le document sous forme de séquences dont la taille est compatible avec la technologie de synthèse. Le résultat est un ensemble de petites séquences (fragments) qui non seulement encode une fraction de l’information d’un document, mais inclut aussi une partie identification et indexation. La partie identification permettra de sélectionner les molécules relatives à un document et la partie indexation permettra de le reconstruire. Le défi ici est que le nombre de nucléotides consacrés à l’identification/indexation reste faible par rapport à la taille totale d’un fragment.
Le codage canal sécurise le signal avec un code correcteur d’erreurs tout en respectant les règles énumérées ci-dessus. En réalité, le processus est plus complexe puisque la séquence engendrée par le code correcteur d’erreurs doit aussi respecter les contraintes biochimiques. Plus globalement, l’enjeu est de mettre au point des mécanismes d’encodage qui supportent les contraintes des molécules d’ADN synthétisées. C’est un sujet difficile, mais qui intéresse un nombre croissant de chercheurs et chercheuses.
Synthèse de l’ADN (étape 2)
La synthèse de l’ADN est un processus purement chimique. Elle consiste à faire croître une chaîne nucléotidique étape par étape via l’incorporation successive des quatre nucléotides (A, C, G ou T). L’approche standard utilise la chimie des phosphoramidites, qui est aujourd’hui entièrement automatisée et mature (visionner cette courte vidéo en anglais qui en explique le principe).
Cependant, l’étape de synthèse représente le principal goulet d’étranglement du stockage sur ADN. Tout d’abord, l’ajout d’une base est une opération très lente, entre 100 et 200 secondes, ce qui se traduit par une vitesse d’écriture des données de l’ordre du kilooctet/jour ! Ensuite, les synthétiseurs ne sont capables de produire que de petits brins d’ADN avec une taille maximum d’environ 300 nucléotides. Enfin, la synthèse chimique utilise des solvants toxiques et peu respectueux de l’environnement.
La parallélisation et la miniaturisation sont les principaux moyens d’augmenter le débit (et aussi de réduire les coûts et l’impact environnemental). En 2022, en utilisant des têtes d’impression miniaturisées, un million de brins d’ADN de 150 nucléotides peuvent être « imprimés » en parallèle sur une période de 24 h.
Depuis les années 2010, une autre technique est sérieusement considérée : la synthèse enzymatique. Elle présente des avantages en termes d’efficacité, de taux de réaction et de respect de l’environnement car elle évite les solvants toxiques et nocifs. L’idée générale est de détourner une ADN polymérase pour écrire un brin d’ADN avec une séquence prescrite. Mais, cette technique est également très lente : les échelles de temps sont semblables à celles de la synthèse chimique.
La synthèse est globalement une procédure avec peu d’erreurs tant que les brins d’ADN à synthétiser ne dépassent pas une certaine longueur. Passé un seuil, le taux d’erreur a tendance à augmenter très fortement. Par conséquent, les tailles des molécules d’ADN de synthèse restent courtes, de l’ordre de 150 à 300 nucléotides.
La synthèse d’ADN reste le maillon faible. Mais beaucoup d’efforts de recherche et d’industrialisation sont engagés pour accroître massivement les débits. On ne peut s’empêcher de mettre en parallèle l’évolution excessivement rapide des technologies de séquençage du début des années 2000. Le premier séquençage du génome humain s’est étalé sur quinze ans (1988-2003), a mobilisé 400 laboratoires et a coûté 3 milliards de dollars. En 2022, le séquençage d’un génome humain ne prend plus que quelques heures et ne coûte que quelques centaines d’euros.
Stockage physique des molécules d’ADN (étape 3)
Le stockage a pour but de conserver les molécules d’ADN dans les meilleures conditions possibles, et sur une très longue période. Cette opération doit tenir compte des spécificités de l’ADN qui, par exemple, se dégrade dès qu’il entre en contact avec des petites molécules organiques, des enzymes, des micro-organismes, de l’oxygène, de l’ozone, etc. L’ADN doit donc être protégé de tous ces agents perturbateurs pour être conservé des milliers d’années. Cette protection peut être réalisée chimiquement ou physiquement.
Le stockage chimique encapsule l’ADN dans des nanoparticules, par exemple des billes de silice. Ces billes sont ensuite stockées dans des plaques à micropuits. Un micropuits stockera, par exemple, l’ensemble des molécules d’ADN relatif à une vidéo.
Le stockage physique considère des capsules de quelques millimètres, comme celles développées par l’entreprise française Imagene, dont l’extérieur est en acier inoxydable et l’intérieur en verre. Leur durée de conservation est estimée à 50 000 ans.
Sélection des molécules d’ADN (étape 4)
D’un point de vue archivage, l’objectif est d’ « encapsuler » de nombreux documents (images, livres, vidéos, etc.) au sein d’un même conteneur. Un document, comme on l’a vu, est encodé sur un très grand nombre de « petits » brins d’ADN. Le conteneur contiendra donc une « soupe » de centaines de milliards de fragments d’ADN correspondant à l’ensemble des documents. Pour accéder à un document unique sans avoir à séquencer l’ensemble du conteneur, son extraction nécessite donc, au préalable, la sélection des brins d’ADN qui lui correspondent.
Contrairement à un disque dur qui localise spatialement et précisément les données relatives à un fichier et qui procure un accès direct et rapide à l’information, les brins d’ADN d’un même document ne sont géographiquement pas regroupés au même endroit. Ils sont dispersés uniformément dans le conteneur. Pour les extraire, il faut donc un mécanisme d’indexation qui puisse les sélectionner de manière unique.
Une stratégie simple repose sur la faculté d’hybridation des brins d’ADN qui a donné lieu à une technologie extrêmement performante : la PCR (polymérase chain reaction). Sans entrer dans les détails, cette technologie amplifie des portions d’ADN en spécifiant un début et une fin par deux courtes séquences de 20 à 25 nucléotides, appelées « amorces ». Si toutes les séquences d’un même document sont encadrées par les mêmes paires d’amorces et qu’on applique une PCR, alors on dupliquera fortement et uniquement les séquences relatives au document recherché. Dans ce contexte, un couple d’amorces se comporte comme l’identifiant d’un document.
D’autres techniques sont possibles pour l’extraction de documents. Cependant, elles demandent toutes des manipulations biotechnologiques qui ont un impact sur l’information stockée dans un conteneur. Elles peuvent en altérer la composition et rendre inaccessible le reste de l’information. À ce stade, il faut prévoir des mécanismes de copie pour éviter une lecture destructrice.
Séquençage de l’ADN (étape 5)
Une molécule d’ADN est décryptée par des appareils nommés séquenceurs. Il existe plusieurs technologies. Toutefois, les plus populaires sont les séquenceurs Illumina, qui appartiennent à la catégorie des séquenceurs de nouvelle génération (années 2000), et les séquenceurs d’Oxford Nanopore Technologies (ONT), qui sont des modèles apparus dix ans plus tard appartenant au groupe des séquenceurs de troisième génération.
La technologie Illumina produit de courts fragments d’ADN, typiquement de 150 à 300 nucléotides issus de la séquence initiale mais dont on a perdu l’enchaînement. Le débit est important grâce à une miniaturisation poussée et une parallélisation massive. La qualité est excellente avec en général moins de 0.5 % d’erreurs.
La technologie ONT, quant à elle, transcrit, au contraire, de longs fragments de plusieurs dizaines de milliers de nucléotides. Il s’agit du plus petit dispositif de séquençage actuellement disponible. Sa portabilité, son prix abordable et la vitesse de production des données en font un outil extrêmement intéressant pour le stockage d’information sur ADN. La qualité du séquençage est cependant moins bonne : le taux d’erreur se situe globalement autour des 5 %.
Le séquençage a un caractère aléatoire. Un séquenceur prend en entrée une solution contenant un grand nombre de molécules d’ADN, mais seule une fraction de ces molécules est transcrite en texte ACGT. Aussi, pour être certain de séquencer toutes les molécules d’ADN au moins une fois, on les duplique fortement avant l’étape de séquençage.
Décodage de l’information (étape 6)
Comme mentionné précédemment, l’étape de séquençage délivre un ensemble de fragments avec une forte redondance. Ces fragments ne sont pas directement transmis au module de décodage. Ils subissent d’abord des traitements inspirés de la bio-informatique pour reconstituer au mieux les textes des séquences. La profondeur de séquençage, c’est-à-dire le nombre de fois en moyenne où un fragment est séquencé, est un paramètre qui conditionne directement la qualité des séquences reconstruites. Après ce traitement, les séquences comportent encore quelques erreurs qui sont normalement corrigées par les codes correcteurs d’erreurs.
Une correction optimisée résulte d’un compromis entre la puissance des codes mis en jeu et l’effort de séquençage. Plus la couverture de séquençage sera importante, meilleure sera la reconstitution des séquences et plus faible sera le taux d’erreur. Dans ce cas, la capacité de correction du code peut être allégée, ce qui implique une redondance moins forte au niveau du signal et donc un espace plus important pour l’information utile. À l’inverse, une faible couverture de séquençage engendrera plus d’erreurs et demandera donc l’élaboration d’un code plus robuste, qui du point de vue de l’information transmise sera moins performant.
En anticipant la diminution exponentielle des coûts de synthèse
Conclusion
Le stockage d’information sur ADN en est encore à ses prémices. Pour l’instant, la phase critique est la phase de synthèse : elle est trop lente et beaucoup trop onéreuse. Mais les années qui viennent pourraient changer la donne grâce à une miniaturisation poussée, un couplage fort avec l’industrie des semi-conducteurs et l’exploitation de nouvelles approches comme la synthèse enzymatique.
De grands acteurs du monde économique considèrent maintenant sérieusement ce moyen de stockage pour l’archivage de grands volumes de données. Le consortium DNA Data Storage Alliance a d’ailleurs été créé en 2020 pour fédérer ce nouveau domaine. Les quatre entreprises fondatrices sont Illumina (séquençage), Microsoft (traitement de l’information), Twist Bioscience (synthèse) et Western Digital (stockage numérique). En 2022, il réunit une cinquantaine de membres de plus de 12 pays différents dont plusieurs universités et instituts de recherche.
Ce qui apparaissait hier encore comme une stratégie de stockage hors de portée, voire du domaine de la science-fiction, devient réalité avec, certes, encore un long chemin à parcourir pour parvenir à une technologie viable et rentable, et conserver sur le long terme le flux croissant d’information que notre société produit. Mais cette ouverture vers un espace de stockage quasi illimité ne peut sans doute pas se passer d’une réflexion plus vaste sur le bon usage de cette nouvelle technologie, et notamment sur la pertinence des données que l’on souhaite archiver sur plusieurs générations.
- Archiver les mégadonnées au-delà de 2040 : la piste de l’ADN. Rapport de l’académie des technologies. 15 octobre 2020.
- Preserving our Digital Legacy: An Introduction to DNA Data Storage. White paper. DNA Storage Alliance. June 2021.
Newsletter
Le responsable de ce traitement est Inria. En saisissant votre adresse mail, vous consentez à recevoir chaque mois une sélection d'articles et à ce que vos données soient collectées et stockées comme décrit dans notre politique de confidentialité
Niveau de lecture
Aidez-nous à évaluer le niveau de lecture de ce document.
Votre choix a été pris en compte. Merci d'avoir estimé le niveau de ce document !

Dominique Lavenier
Directeur de recherche CNRS à l’Institut de recherche en informatique et systèmes aléatoires (IRISA), membre de l'équipe de recherche GENSCALE.

Marc Antonini
Chercheur CNRS au Laboratoire d'informatique, signaux et systèmes de Sophia Antipolis (I3S).

Anthony Genot
Chercheur CNRS dans l’unité LIMMS (Laboratory for Integrated Micro Mechatronics Systems) à l’Université de Tokyo.